目录
TCGA官网操作
首先打开TCGA官网(The Cancer Genome Atlas)GDC(Genomic Data Commons)data portal 2.0

左上角是 GDC Data Portal 徽标,它链接到 GDC Data Portal 的主页。徽标下方是按以下顺序排列的链接:
• 分析中心:用于访问 GDC 数据门户中所有工具的中心枢纽;
• 项目:允许浏览 GDC 数据门户中的所有项目;
• 队列生成器:队列生成器工具由各种临床和生物样本过滤器组成,用于构建用于分析的自定义队列;
• 存储库:允许浏览与队列关联的文件。
对于分析中心(Analysis center),网站提供了很多方便的分析功能,大家可以自行摸索。尤其对于单个基因或者少数基因数据的获取可以基于gene expression clustering获得,不用下载完整的数据。

使用数据分析和数据存储库之前需要通过队列生成器(cohort builder)创建自己感兴趣的cohort,比如TCGA-UCEC,选好之后自定义命名:
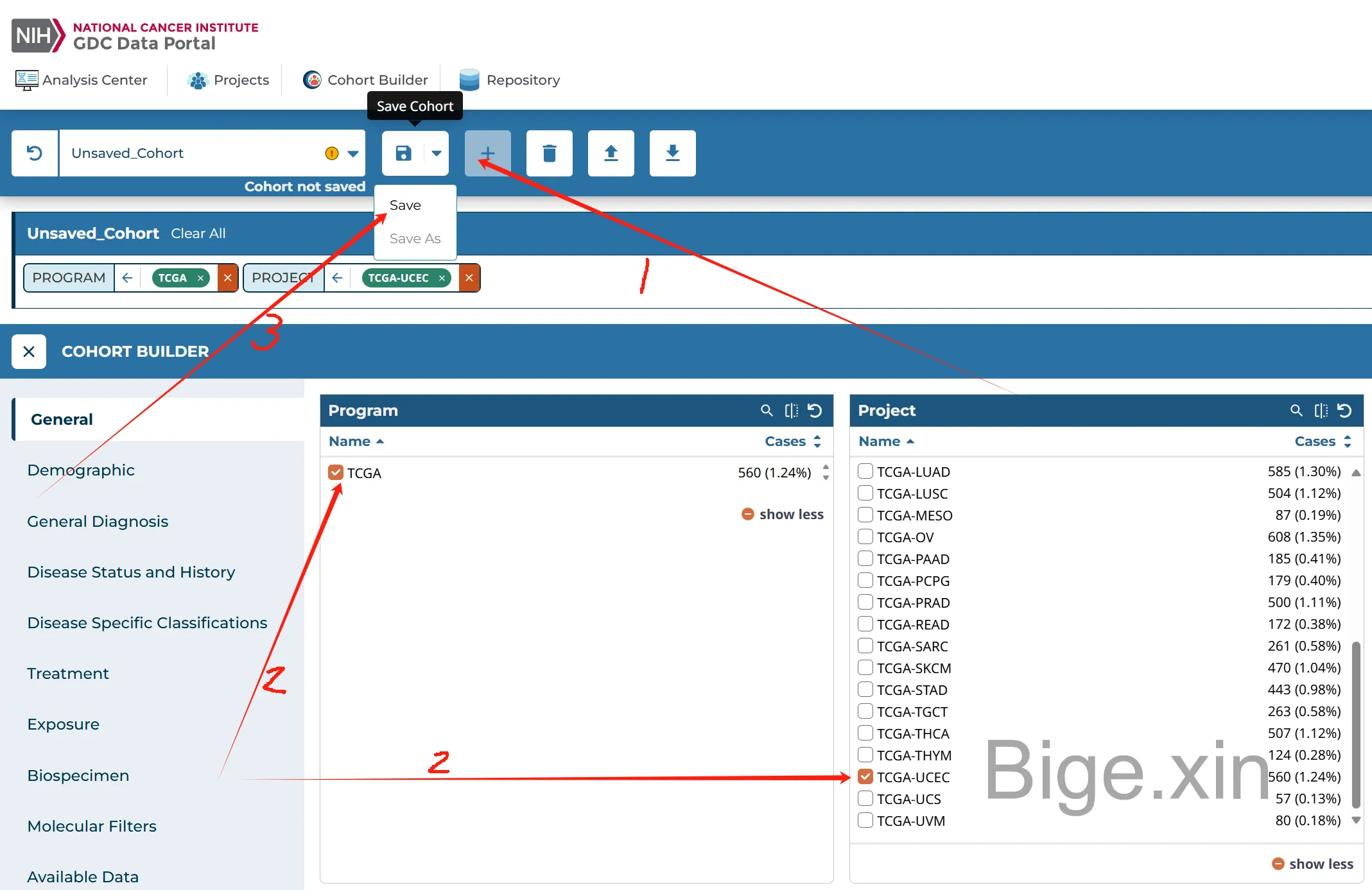
转到存储库(Repository),选择自己的cohort,选择自己需要的数据类型,add all files to cart:
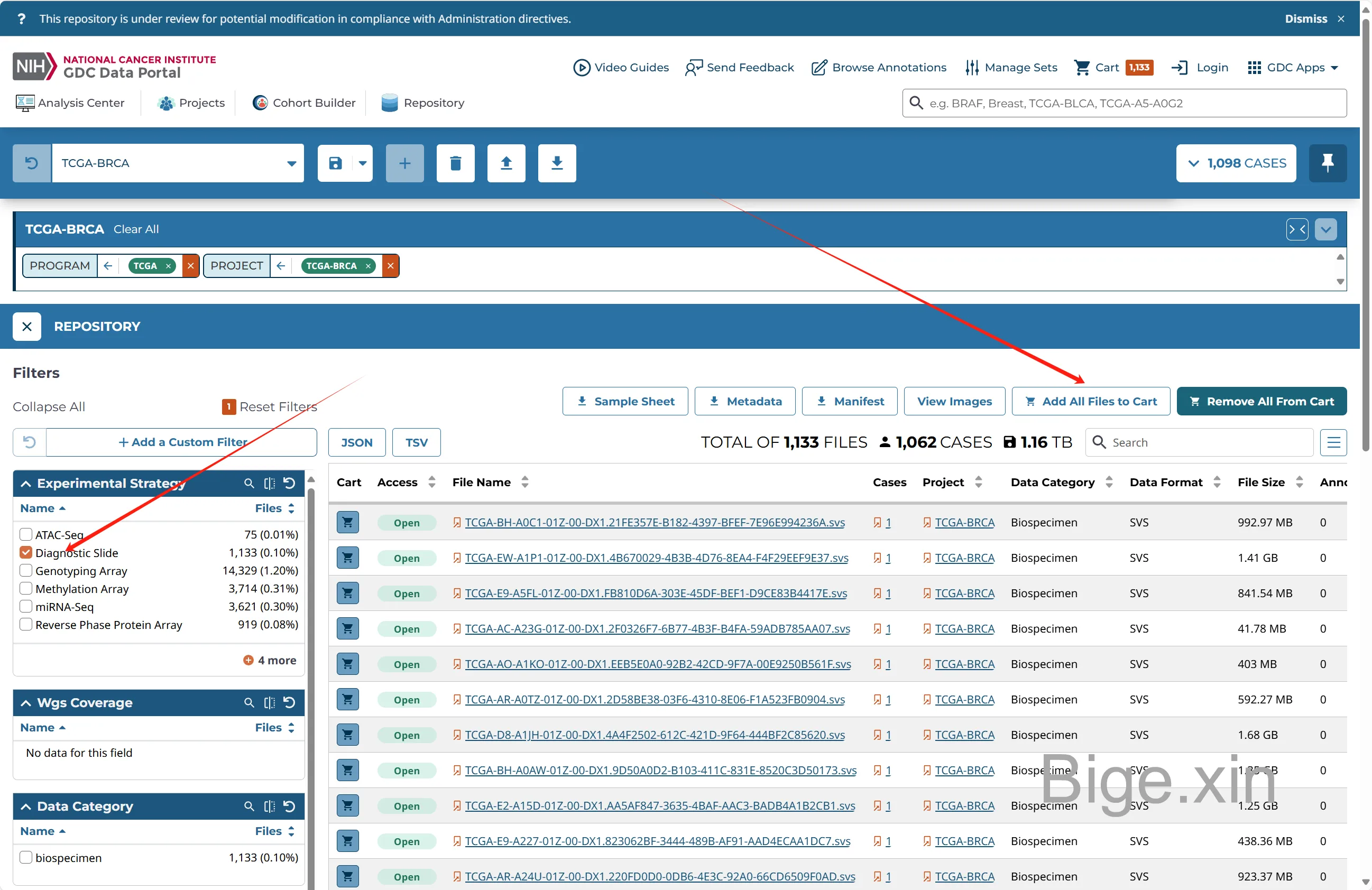
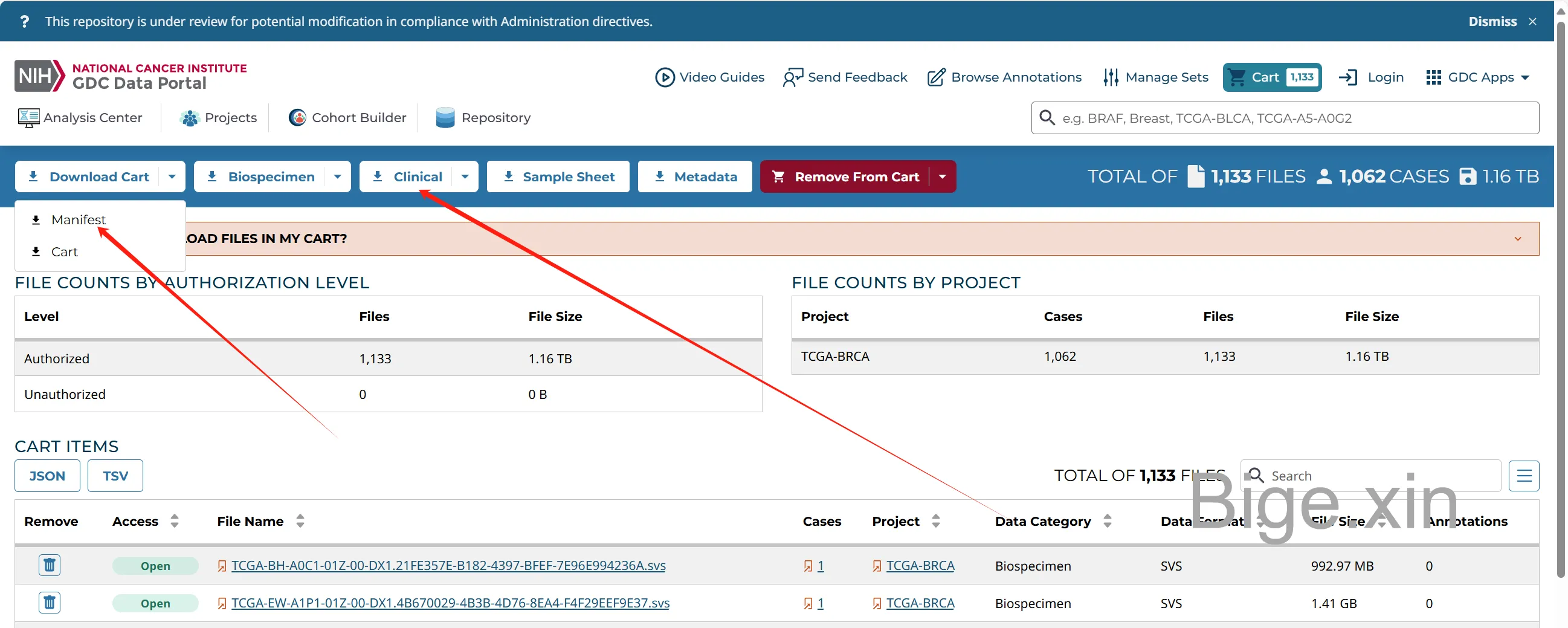
最后,进入cart下载数据,一般下载Manifest文件和临床文件clinic data即可,按需下载。
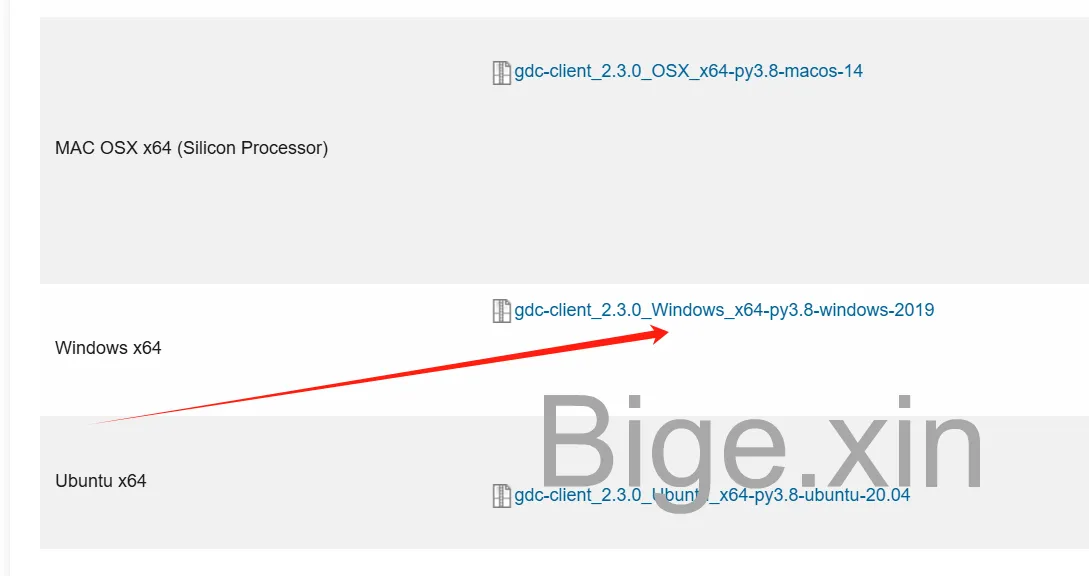
下载TCGA官网推荐下载工具GDC,windows用户,下载解压后是一个gdc-client.exe文件。
本地电脑操作
在电脑上新建一个文件夹,比如E:\TCGA-BRCA用来盛放下载的数据,将刚刚下载的Manifest文件和gdc-client.exe文件放进该文件夹,然后在该文件夹中继续新建gdc-client.dtt文件,用记事本编辑保存下面内容:
[download] no_auto_retry = False no_file_md5sum = False save_interval = 1073741824 retry_amount = 100 n_processes = 20 no_annotations = False no_related_files = False http_chunk_size = 4096 no_segment_md5sum = False server = https://api.gdc.cancer.gov wait_time = 100.0 no_verify = True dir = .
上面代码是下载工具的参数配置信息,用逻辑分析一下就知道怎么配置了,主要可以配置的是http_chunk_size、wait_time、retry_amount、no_verify、n_processes 解释一下:
- http_chunk_size:这是每一次http请求的数据块大小,如果越大,等待时间越长,可能就会超时,就pa断了,所以为了保证每一次请求数据块都能顺利到达本地,就调小一点(这一步相当关键了)
- wait_time:http请求的等待时间,如果超过这个等待时间,也会pa断了,所以这一步就可以调大一丢丢
- retry_amount:重试次数,比如说超过等待时间,就会再重新尝试访问,尝试访问次数超过,pa断了,所以这一步也可以调大一丢丢
- no_verify:不做校验,(不知道它底层用什么做校验的),为了求速度,不做校验(想保证数据完整性就不要改这个啦)
- n_processes:配置和CPU大小一致就行了
最后快捷键win+R,输入cmd回车进入,输入下面代码,即可开启愉快的下载历程啦!
C:\Users\liudh>E: E:\TCGA-BRCA>gdc-client.exe download --config gdc-client.dtt -m gdc_manifest.txt


本文作者:比格心
本文链接:
版权声明:本博客所有文章除特别声明外,均采用 BY-NC-SA 许可协议。转载请注明出处!
